1. 研究借助于高通量筛选技术和计算生物学手段,对58000多种转录因子组合是否可协同结合DNA进行了系统性评估,鉴定出2198对协同作用的转录因子,并解析了协同作用转录因子识别的DNA基序(图a)。进一步深入研究发现,转录因子间的协同作用目前还不能被AlphaFold等人工智能工具所预测(图b)。因而,研究提供的迄今为止最全面的转录因子协同作用数据库,以及协同作用转录因子识别的DNA基序库,为科学家们深入探索转录因子的各种生物学功能提供了重要的数据支撑和理论依据。研究团队还发现,协同作用致使800多对转录因子识别的DNA基序发生改变。单细胞数据分析和基因编辑小鼠实验证实,协同转录因子识别的新型DNA序列可驱动基因的细胞特异性表达,在发育早期细胞命运决定中发挥至关重要的作用(图c)。
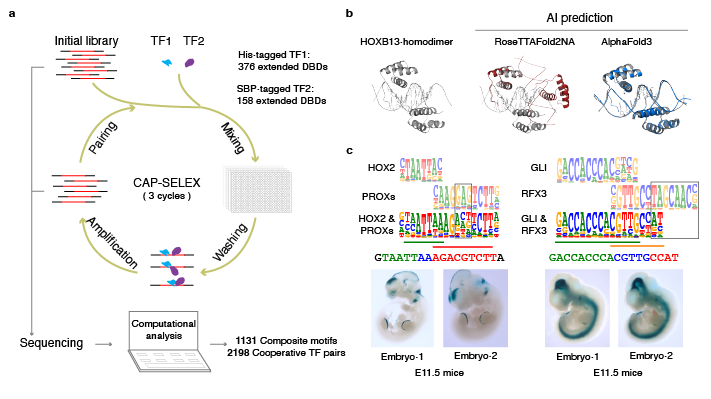
图:(a) 高通量CAP-SELEX技术原理图;(b) 协同作用转录因子复合体结构(左)和人工智能工具预测的结构(右);(c) 协同作用转录因子识别新型调控密码(上),调控基因细胞特异性表达(下)。
综上所述,本研究从更高的维度上拓展了人类基因组的调控密码,证实了协同作用转录因子通过解读这些新型调控密码,指导基因的细胞特异性表达,调控早期胚胎发育等高度时空特异的生物学过程,该成果于2025年发表于Nature (https://doi.org/10.1038/s41586-025-08844-z)。
2. 基于nanopore 三代测序技术,我们开发了一种可在单分子水平同时检测DNA甲基化和组蛋白修饰的nanoHiMe-seq方法,探索了DNA甲基化与常染色质区域H3K4me3修饰、以及异染色质区域H3K27me3修饰的内在联系,揭示了人类细胞内广泛存在的父源和母源染色质表观修饰的不平衡性。nanoHiMe-seq方法为深入探索细胞命运决定过程中表观修饰图谱建立的分子机制提供了一种有力工具,该成果于2022年底发表于Nature Commun (https://www.nature.com/articles/s41467-022-35650-2)。
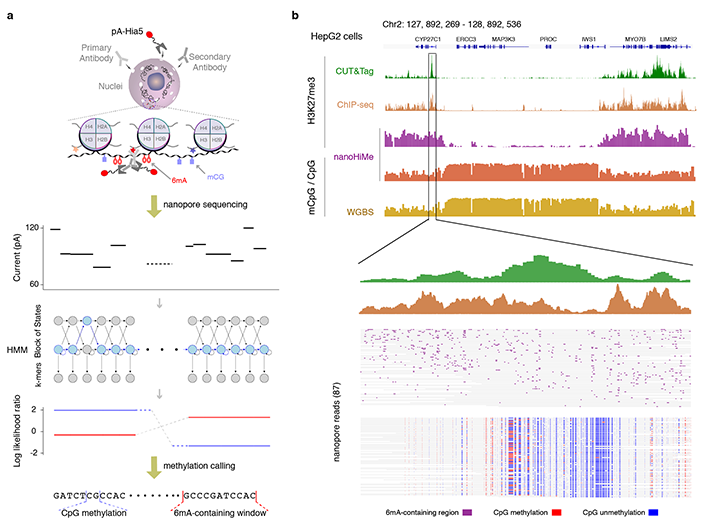
图:nanoHiMe-seq方法(a)在单分子水平同时检测DNA甲基化和组蛋白H3K27me3修饰(b)。
3. 基于Illumina测序技术,我们建立了一种叫methylation-sensitive systematic evolution of ligands by exponential enrichment(SELEX)的方法,系统性解析转录因子结合DNA的序列特异性、并评估DNA的表观遗传修饰对二者结合的影响。运用该方法,成功地为542个人类转录因子建立了DNA序列特异性模型,并首次在全基因组水平揭示了CpG甲基化对转录因子结合DNA的序列特异性的影响,发现了175个与胚胎发育和器官发生相关的转录因子,如重编程转录因子OCT4和人类躯体发育相关的HOX家族成员 HOX9-13等,更倾向于结合CpG甲基化DNA序列。体内研究发现某些喜欢结合甲基化DNA的转录因子与细胞重编程过程中基因组特定位点的甲基化状态改变有关,研究成果已在Science (https://www.science.org/doi/10.1126/science.aaj2239)杂志发表。
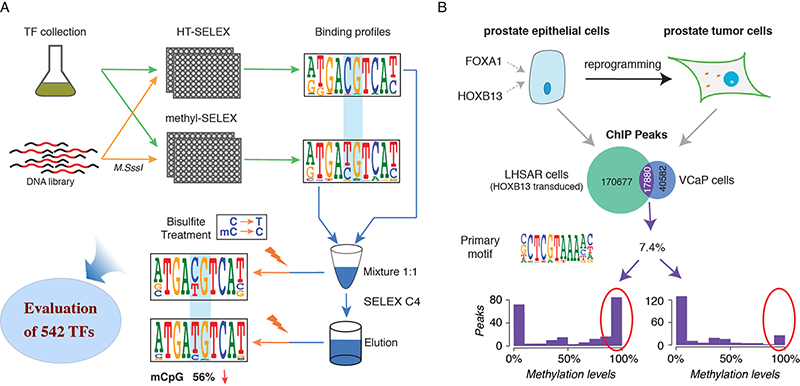
图:A. Methyl-sensitive SELEX分析转录因子识别的DNA序列以及DNA甲基化影响;B. 转录因子HOXB13诱导DNA去甲基化。